使用BioEdit生成和绘制蛋白质进化树 虽然方法简单、上手快,但是由于 BioEdit 功能有限,使用起来很不方便。如果经常进行类似分析,那还是要使用 MEGA 6 这一个系统进化分析的“大杀器”。
第一步:下载和安装 MEGA 6。各平台均有发行版,在此不再赘述。
第二步:多序列比对,这是构建系统进化树的必要条件。
点击第一个按钮,选择 Edit/Build Alignment。
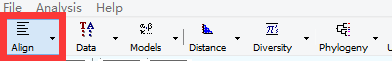
正如题目所说的,我们需要选择一个含有多个序列的 Fasta 文件。
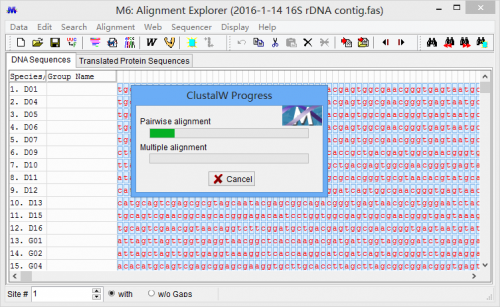
从菜单选择 Alignment – Align By xxx 如果序列比较多,那比对需要花上一些时间。
第三步:进化分析。
在比对完成的窗口中直接选择 Phylogenetic Analysis。
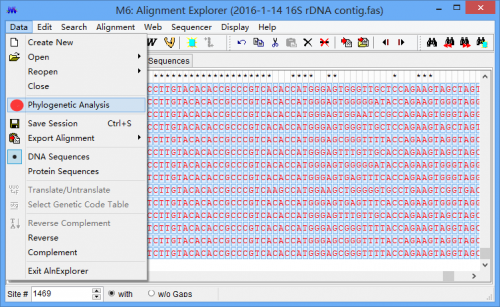
第四步:构建进化树。
在主窗口页面上点击 Phylogeny 这个按钮,选择一种方法构建进化树,
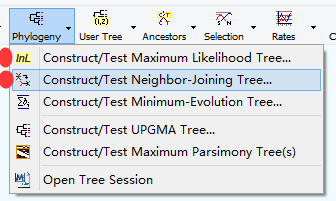
点击 Compute 开始计算。
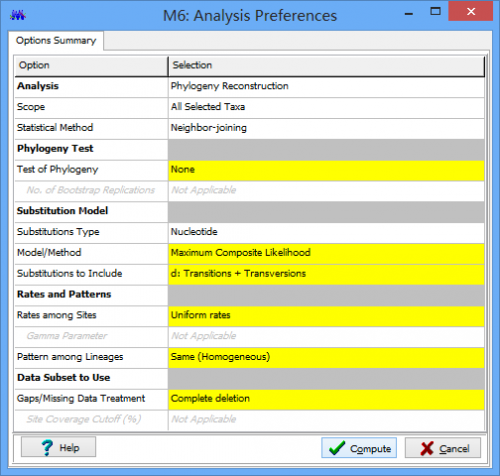
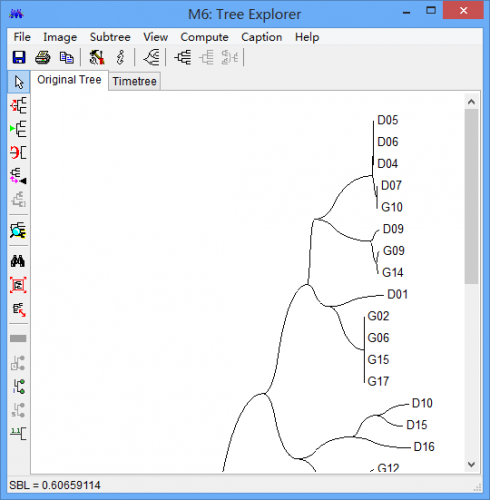
现在你将得到你所需要的进化树了。